 Schneller zu neuen Wirkstoffen durch Genomik, Bioinformatik und chemische Analytik
Schneller zu neuen Wirkstoffen durch Genomik, Bioinformatik und chemische AnalytikHubertus von Bramnitz
Frankfurt am Main (Weltexpresso) - Viele wichtige Wirkstoffe wie Antibiotika, Immunsuppressiva oder Mittel gegen Krebs stammen aus dem Stoffwechsel von Mikroorganismen. Diese produzieren oft ganze Stoffgemische, deren natürliche Funktion bisher kaum bekannt ist. Auf der Suche nach natürlichen Vorbildern für neue Arzneistoffe haben Biotechnologen und Bioinformatiker der Goethe-Universität nun zusammen die Stoffgemische zweier verwandter Bakterienarten untersucht.
„Wir vermuten, dass die Produktion von Naturstoffen für das Leben von Mikroorganismen wie Bakterien oder Pilzen wichtige Funktionen besitzen. Wenn wir diese verstehen würden, gelänge es vermutlich häufiger, eine verwandte Anwendung für diese Substanzen zu finden. So käme man auch zu neuartigen Medikamenten“, erläutert Prof. Helge Bode, Merck Stiftungsprofessur für Molekulare Biotechnologie an der Goethe-Universität, den Forschungsansatz.
Die Gruppe von Bode untersucht schon seit einigen Jahren die Funktion von Bakterien der Gattung Photorhabdus und Xenorhabdus. Sie sind ideale Modellsysteme für die Analyse der natürlichen Funktion von Naturstoffen, da sie viele verschiedene Naturstoffe produzieren und sowohl als Symbionten im Darm von Fadenwürmern leben als auch zusammen mit diesen Insektenlarven befallen und töten können. Die Bakterien müssen also in der Lage sein, verschiedene höhere Lebewesen zu unterscheiden: Mit bestimmten Naturstoffen fördern sie das Wachstum der Fadenwürmer, mit denen sie zusammenleben, und mit anderen töten sie Insekten.
Um zu verstehen, wie diese Fähigkeiten im Laufe der Evolution entstanden sind, hat Bode mit der Gruppe von Prof. Ingo Ebersberger, Bioinformatiker an der Goethe-Universität, zusammen gearbeitet. Er hat die bakteriellen Genome von 30 Bakterienarten mit bioinformatischen Methoden analysiert. Dank der Kombination mit modernster chemischer Analytik konnten die beiden Gruppen schneller neue Naturstoffe der Bakteriengattungen Photorhabdus und Xenorhabdus identifizieren.
Die vergleichende Analyse erlaubte zudem eine Identifizierung von Naturstoffen, die in Untergruppen oder auch fast in allen Arten vorkommen. Das lässt vermuten, dass sie eine wichtige Rolle im Lebenszyklus dieser Bakterienarten spielen.
„Die in unserer Publikation genutzten Technologien erlauben zwar die Identifizierung von biologisch relevanten Mustern, diese in ihrer Bedeutung im Detail zu verstehen, ist jedoch noch ein Stück Arbeit“, erklärt Bode. Dieser Aufgabe wird er sich mit seiner Gruppe in den nächsten Jahren widmen.
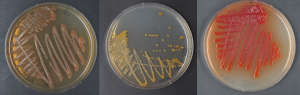
Foto: Wachstum ausgewählter Xenorhabdus (links und mitte) und Photorhabdus (rechts) Kolonien auf Agarplatten. Die unterschiedliche Pigmentierung weist bereits direkt auf die Produktion verschiedener Naturstoff-Pigmente hin. Fotos: AK Bode © uni-frankfurt.de
Info:
Publikation:
Nicholas J. Tobias, Hendrik Wolff, Bardya Djahanschiri, Florian Grundmann, Max Kronenwerth, Yi-Ming Shi, Svenja Simonyi, Peter Grün, David Shapiro-Ilan, Sacha J. Pidot, Timothy P. Stinear, Ingo Ebersberger and Helge B. Bode: Natural product diversity associated with the nematode symbionts Photorhabdus and Xenorhabdus, Nature Microbiology, www.nature.com/naturemicrobiology, DOI: 10.1038/s41564-017-0039-9

