 Neue Methode ermöglicht detaillierte Analyse der Zielmoleküle
Neue Methode ermöglicht detaillierte Analyse der Zielmoleküle
Susanne Sonntag
Frankfurt am Main (Weltexpresso) - Ubiquitin ist ein kleines Molekül, das im Körper an andere Proteine angehängt wird und so deren Funktion kontrollieren und verändern kann. Die Anheftung erfolgt auf viele verschiedene Arten, entweder einzeln oder in verzweigten Ketten aus mehreren Ubiquitin-Molekülen.
Dadurch entstehen sehr unterschiedliche Strukturen, die wiederum verschiedenste Effekte in Zellen erzielen können. Wissenschaftler sprechen mittlerweile von einem regelrechten Geheimcode. Um diesen zu entschlüsseln, haben Forscher der Goethe-Universität in Zusammenarbeit mit Kollegen von der Universität von Tübingen, der Queen Mary Universität und dem Francis Crick Institut in London eine neue Methode entwickelt.
Erst vor wenigen Jahren entdeckten Forscher, dass Ubiquitin nicht nur verzweigte, sondern auch lineare Ketten bilden kann, bei denen jeweils der Anfang eines Ubiquitin-Moleküls an das Ende eines anderen geknüpft wird. Bislang sind erst zwei Enzyme bekannt, die den Auf- und Abbau solcher linearer Ubiquitin-Ketten regulieren. Beide werden im Institut für Biochemie II der Goethe-Universität intensiv erforscht. Welche Zielproteine mit linearen Ubiquitin-Ketten modifiziert werden, und warum, blieb jedoch weitestgehend unklar. Die von dem Forscherteam um Koraljka Husnjak von der Goethe-Universität Frankfurt neu entwickelte Methode ermöglicht nun die systematische Analyse dieses speziellen Kettentypus.
„Der bislang vergleichsweise schleppende Fortschritt war vor allem darauf zurückzuführen, dass es keine geeigneten Methoden gab, um mit linearen Ubiquitin-Ketten modifizierte Proteine spezifisch im Massenspektrometer zu erfassen“, erklärt die gebürtige Kroatin Husnjak. Ihr Team hat das Problem gelöst, indem es das Ubiquitin-Molekül intern so veränderte, dass es einerseits innerhalb der Zelle funktionstüchtig bleibt, andererseits aber bei einer Analyse von Proteingemischen im Massenspektrometer erkannt werden kann.
Künftig lässt sich also genau nachweisen, welche Zielproteine durch lineares Ubiquitin verändert werden und an welcher Position des Proteins die entsprechende Modifikation sitzt. Die Wissenschaftler bewerten diesen neuen, äußerst sensitiven Ansatz als einen wichtigen Durchbruch, der zu einem bedeutend besseren Verständnis der Funktion der linearen Ubiquitinierung und ihrer Rolle bei Erkrankungen führen wird.
Dr. Husnjak konnte so bereits mehrere Proteine identifizieren, von denen bislang nicht bekannt war, dass sie linear ubiquitiniert werden. Unter anderem entdeckte sie neue essentielle Komponenten innerhalb eines Signalweges, der bei Entzündungen zentral ist. „Die durch lineare Ubiquitin-Ketten übertragenen Signale spielen zum Beispiel eine wichtige Rolle bei der Regulation von Immunantworten, der Abwehr von Infektionen und bei immunologischen Erkrankungen. Bislang wissen wir nur ansatzweise, wie aus kleinen Fehlern in diesem System schwere Krankheiten entstehen und wie man gezielt therapeutisch eingreifen kann“, so die Forscherin über das Potential der neuen Methode.
Fehler im Ubiquitin-System werden mit zahlreichen Erkrankungen in Zusammenhang gebracht, so zum Beispiel mit der Entstehung von Krebs, mit neurodegenerativen Erkrankungen wie Parkinson, aber auch mit dem Verlauf von Infektionen und Entzündungen.
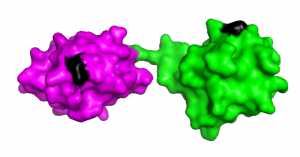
Foto: Schematische Darstellung von zwei linear verknüpften Ubiquitin-Molekülen. Die interne Markierungsstelle ist schwarz markiert (c) Koraljka Husnjak, erstellt mit PyMOL Software
Info:
Publikation:
Katarzyna Kliza, Christoph Taumer, Irene Pinzuti, Mirita Franz-Wachtel, Simone Kunzelmann, Benjamin Stieglitz, Boris Macek & Koraljka Husnjak. Internally tagged ubiquitin: a tool to identify linear polyubiquitin-modified proteins by mass spectrometry. Nature Methods 2017. doi:10.1038/nmeth.4228

