 Struktur zur Verbreitung von Resistenzgenen entschlüsselt/ Publikation in eLife
Struktur zur Verbreitung von Resistenzgenen entschlüsselt/ Publikation in eLife
Hubertus von Bramnitz
Frankfurt am Main (Weltexpresso) - Multiresistente pathogene Keime entstehen durch den Transfer von Resistenzgenen und Virulenzfaktoren von einem Bakterium auf ein anderes. Der wichtigste Mechanismus ist dabei der Transfer freier DNA ohne Zell-Zell-Kontakt, da dieser auch über die Artgrenzen hinweg erfolgt. Haben Sie verstanden? Hilft das Foto?
Wissenschaftlerinnen der Goethe-Universität und des Max-Planck-Instituts für Biophysik haben nun die Vermutung bestätigt, dass lange Härchen-artige Strukturen für den DNA-Transfer verantwortlich sind. Wie die Forscherinnen und Forscher in der aktuellen Ausgabe der Fachzeitschrift eLife berichten, konnten sie den Mechanismus mithilfe der Cryo-Tomographie auf molekularer Ebene aufklären.
Umfangreiche genetische und biochemische Analysen hatten in den letzten Jahren eine ungeahnte Komplexität des Vorgangs und der daran beteiligten Transportmaschinerie gezeigt. „Seit einiger Zeit kennen wir die beteiligten Proteine und haben auch Strukturen einzelner Komponenten, aber wir konnten nur vermuten, wo sie sich in der Zelle befinden und wie sie in der Bakterienmembran räumlich zueinander angeordnet sind“, erklärt Prof. Beate Averhoff von der Abteilung Molekulare Mikrobiologie & Bionergetik der Goethe Universität.
„All unsere Daten ließen nur eine Schlussfolgerung zu: dass an der DNA-Aufnahme Härchen der Bakterien beteiligt sind, die in der Cytoplasmamembran ihren Anfang nehmen und mittels hochkomplexer Portale nach außen in die Umgebung geleitet werden.“ Diese Härchen oder Pilus-artigen Strukturen durchspannen die gesamte Zellperipherie und reichen mit einer Länge, die das Vielfache einer Bakterie erreichen kann, ins Ökosystem.
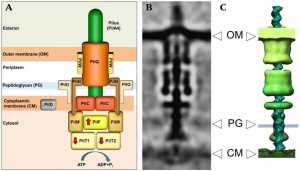
Im Modelorganismus Thermus thermophilus hat Beate Averhoff zusammen mit Vicki Gold und Werner Kühlbrandt vom MPI für Biophysik die Struktur des Typ-IV-Pilus und des Portals dieses DNA-Transporters mithilfe der Cryo-Tomographie erstmals sichtbar gemacht. Diese neue und enorm leistungsfähige Methode wird in der Abteilung von Prof. Werner Kühlbrandt zur Aufklärung der Struktur makromolekularer Komplexe eingesetzt. Der detektierte DNA-Transporter durchspannt die gesamte Zellperipherie und ist circa 35 Nanometer lang und 15 Nanometer breit.
Für die Forscherinnen hochinteressant war die Beobachtung, dass der Komplex in zwei Konformationen vorliegt, einer geschlossenen und einer offenen. Dabei fanden sie zwei Portale, die sich entweder beide gleichzeitig öffnen oder schließen. Die koordinierte Dynamik dieser zwei Portale ist entscheidend für den Durchtritt der Pili und somit für die Aufnahme freier DNA aus der Umgebung. Durch das Öffnen und Schließen der beiden Portale ändert sich auch die räumliche Struktur weiter entfernter Proteine, darunter die der Kraftwerke des DNA-Translokators, welche die Energie für den Transport der DNA bereitstellen.
Gelingt es, die Struktur des DNA-Translokators in der Zellmembran aufzuklären, wird es künftig möglich sein, die räumliche Anordnung der einzelnen Proteine, ihre Interaktion und ihre Rolle im DNA-Transfer zu analysieren. „Dann ist die Entwicklung von Hemmstoffen zur Inhibition des DNA-Transfers und damit zur Verhinderung der Ausbreitung von Resistenzgenen in greifbare Nähe gerückt“, prognostiziert Averhoff. Die Fragestellung sei außerdem relevant vor dem Hintergrund der Freisetzung gentechnisch veränderter Organismen.
Publikation:
Vicki A M Gold, Ralf Salzer, Beate Averhoff und Werner Ku?hlbrandt. Structure of a type IV pilus machinery in the open and closed state. eLife 2015;10.7554/eLife.07380
Foto:
Typ IV Pilus-Maschinerie und seine Funktion als DNA-Transporter: Schematisches Modell (A), Durchschnitt der Subtomogramme (B) und resultierende 3D Oberfläche (C) im geöffneten Zustand.
Informationen: Prof. Beate Averhoff, Molekulare Mikrobiologie und Bioenergetik, Institut für Molekulare Biowissenschaften, Campus Riedberg, (069)-798-29509; Diese E-Mail-Adresse ist vor Spambots geschützt! Zur Anzeige muss JavaScript eingeschaltet sein!.
Internet: www.uni-frankfurt.de

