 Fluoreszierende Proteinmarker unter Hochdruck eingeschleust
Fluoreszierende Proteinmarker unter Hochdruck eingeschleust
Hubertus von Bramnitz
Frankfurt am Main (Weltexpresso) - Bestimmte Proteine in der Zelle aufzuspüren, gleicht der Suche nach einer Nadel im Heuhaufen. Um Proteine orten und ihre Funktion in der lebenden Zelle entschlüsseln zu können, versuchen Forscher, sie mit fluoreszierenden Molekülen zu markieren. Doch diese lassen sich oft nicht in genügender Anzahl einschleusen.
Eine Forschergruppe der Goethe Universität fand jetzt in Kooperation mit amerikanischen Kollegen eine Lösung für dieses Problem. In der aktuellen Ausgabe der Fachzeitschrift Nature Communications berichten sie über ein Verfahren, mit dem sie chemische Sonden unter Druck fein dosiert in lebende Zellen einschleusen können.
„Obwohl immer mehr Proteinmarkierungsmethoden synthetische Fluoreszenzfarbstoffe nutzen, leiden diese oft unter Problemen wie Zellpermeabilität und geringer Markierungseffizienz. Auch kann man sie nur eingeschränkt mit anderen Proteinmarkierungsmethoden verwenden“, erklärt Dr. Ralph Wieneke vom Institut für Biochemie der Goethe-Universität.
Die Arbeitsgruppe um Wieneke und Prof. Robert Tampé hat schon vor längerer Zeit einen Marker entwickelt, der ausgewählte Proteine mit einer Präzision von wenigen Nanometern in der Zelle lokalisiert. Es handelt sich um ein hochspezifisches Schlüssel-Schloss-Paar, bestehend aus dem kleinen synthetischen Molekül trisNTA und einem genetisch codierten His-tag.
Um diesen Proteinmarker in die Zellen zu schleusen, verwendeten die Frankfurter Forscher zusammen mit Kollegen des Massachussetts Institute of Technology (MIT), Cambridge, USA, ein Verfahren, bei dem der Marker zusammen mit den Zellen in Lösung schwimmt. Diese wird dann durch verengte Kanäle gepresst (cell squeezing). Unter dem Druck nehmen die Zellen die fluoreszierenden Sonden mit einer Effizienz von über 80 Prozent auf. Das Hochdurchsatz-Verfahren erlaubt es, eine Million Zellen pro Sekunde durch die künstliche Kapillare zu pressen.
Da der Marker in hohem Maße spezifisch an die gewünschten Proteine bindet und sich seine Konzentration in der Zelle präzise regulieren ließ, konnten die Forscher hochauflösende mikroskopische Bilder von lebenden Zellen aufnehmen. Zudem konnten sie den Marker erst an sein Zielmolekül binden lassen, wenn es durch Licht aktiviert wurde. So lassen sich zelluläre Prozesse nicht nur räumlich, sondern auch zeitlich präzise verfolgen.
Die Forscher konnten ihre Markierungsmethode in lebenden Zellen sogar mit anderen Proteinmarkierungsmethoden kombinieren. So wird es möglich, gleichzeitig mehrere Proteine in Echtzeit zu beobachten. „Wir konnten eine Vielzahl an fluoreszenzmarkierten trisNTAs mittels cell squeezing in Zellen einschleusen. Damit erweitern sich die Möglichkeiten der konventionellen und der hochauflösenden Lebendzellmikroskopie ungemein“, erklärt Prof. Robert Tampé. Zukünftig wird man dynamische Prozesse in lebenden Zellen mit höchster Auslösung in Zeit und Raum verfolgen können.
Foto: © Universität Frankfurt.
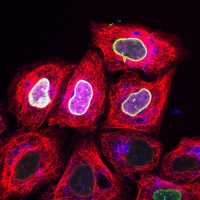
Mit Hilfe des Schlüssel-Schloss-Prinzips lässt sich selektiv das Kernhüllenprotein Lamin A mit der fluoreszenzmarkierten Sonde trisNTA (grün) anfärben. Simultan können in der gleichen Zelle durch orthogonale Markierungsmethoden weitere Proteine visualisiert werden (Histon2B in mangenta; Lysosomen in blau; Mikrotubuli in rot).
Info:
Publikation: Alina Kollmannsperger, Armon Sharei, Anika Raulf, Mike Heilemann, Robert Langer, Klavs F. Jensen, Ralph Wieneke & Robert Tampé: Live-cell protein labelling with nanometre precision by cell squeezing, in: Nature Communications, 7:10372,
DOI: 10.1038/ncomms10372
www.nature.com/naturecommunications
Informationen: Dr. Ralph Wieneke, Institut für Biochemie, Campus Riedberg, Tel.: (069) 798-29477, Diese E-Mail-Adresse ist vor Spambots geschützt! Zur Anzeige muss JavaScript eingeschaltet sein!.

